高性能計算システム研究部門の高橋大介教授が、IEEE Computer Society の 2023 Class of IEEE Computer Society Distinguished Contributors に選出されました。
関連リンク:https://www.computer.org/membership/distinguished-contributors
高性能計算システム研究部門の高橋大介教授が、IEEE Computer Society の 2023 Class of IEEE Computer Society Distinguished Contributors に選出されました。
関連リンク:https://www.computer.org/membership/distinguished-contributors
生命科学研究部門の原田隆平准教授が、公益財団法人UBE学術振興財団の第64回学術奨励賞を受賞しました。
受賞研究課題は、「疾患原因となる分子間相互作用を特定する異常検知システムの開発(医学)」です。
関連リンク:https://www.ube.co.jp/ube_s_zaidan/grants.html#y2023
The Joint Center for Advanced High Performance Computing (JCAHPC), which is jointly operated under the collaboration between the Center for Computational Sciences, University of Tsukuba, and the Information Technology Center, the University of Tokyo, has named its new supercomputer system “Miyabi,” which is planned to start its entire operation in January 2025.
See the News article on JCAHPC website for details.
筑波大学計算科学研究センターと東京大学情報基盤センターが共同運営する、最先端共同HPC基盤施設(JCAHPC:Joint Center for Advanced High Performance Computing、施設長:朴泰祐)は、2025年1月に稼働を開始する次期スーパーコンピュータシステムの名称を “Miyabi”(みやび)に決定しました。
詳細はJCAHPCのニュース記事をご覧ください。
*This colloquium has been canceled.
Title: Climate Change Research: Insights from KAUST
Speaker: Prof Ibrahim Hoteit(King Abdullah University of Science and Technology)
Date: 2 April 2024
Time: 13:30-14:15
Venue: Center for Computational Sciences, Workshop Room
Language: English
*After the invited presentation, the following seminar is scheduled.
13:30-14:15
Climate Change Research: Insights from KAUST
Prof Ibrahim Hoteit, King Abdullah University of Science and Technology
14:15-14:45
Urban Climate Modeling for urban and district scales: WRF-UCM and City-LES
Prof Hiroyuki Kusaka, University of Tsukuba
14:45-15:15
Urban Extreme Weather and Climate Change
Asst Prof Quang-Van Doan, University of Tsukuba
15:1515:30
General Discussion

*第140回計算科学コロキウムは中止となりました(2024.04.01追記)*
第140回計算科学コロキウムを開催いたします。多数のご来聴をお待ちしております。
講演タイトル:Climate Change Research: Insights from KAUST
講演者: Prof Ibrahim Hoteit(King Abdullah University of Science and Technology)
日時: 2024年4月2日(火) 13:30-14:15
場所: 計算科学研究センター ワークショップ室
言語: English
*計算科学コロキウムに続けて、セミナーが開催されます。
全体のスケジュールは以下の通りです。
13:30-14:15
Climate Change Research: Insights from KAUST
Prof Ibrahim Hoteit, King Abdullah University of Science and Technology
14:15-14:45
Urban Climate Modeling for urban and district scales: WRF-UCM and City-LES
Prof Hiroyuki Kusaka, University of Tsukuba
14:45-15:15
Urban Extreme Weather and Climate Change
Asst Prof Quang-Van Doan, University of Tsukuba
15:1515:30
General Discussion

筑波大学計算科学研究センターでは、東京大学情報基盤センターが運用する高性能メニーコアクラスタ Wisteria-O(7,680ノード、A64FX、25.9 PFLOPS)及び筑波大学計算科学研究センターが運用するビッグメモリスーパコンピュータPegasus (120ノード、 SPR、 H100、不揮発性メモリ、 6.5 PFLOPS)とGPU, FPGA 混載型クラスタCygnus (78ノード、 V100、 FPGA、2.3 PFLOPS)の3台のスーパコンピュータについて、各システムの20%(Wisteria-Oについては筑波大割当分の20%)を目安とした計算機資源を、全国共同利用機関として有償の一般利用に供することといたします。
2024年度の一般利用を募集しますので、希望される方は以下の要領でご応募下さい。
一般利用
Feb 22, 2024
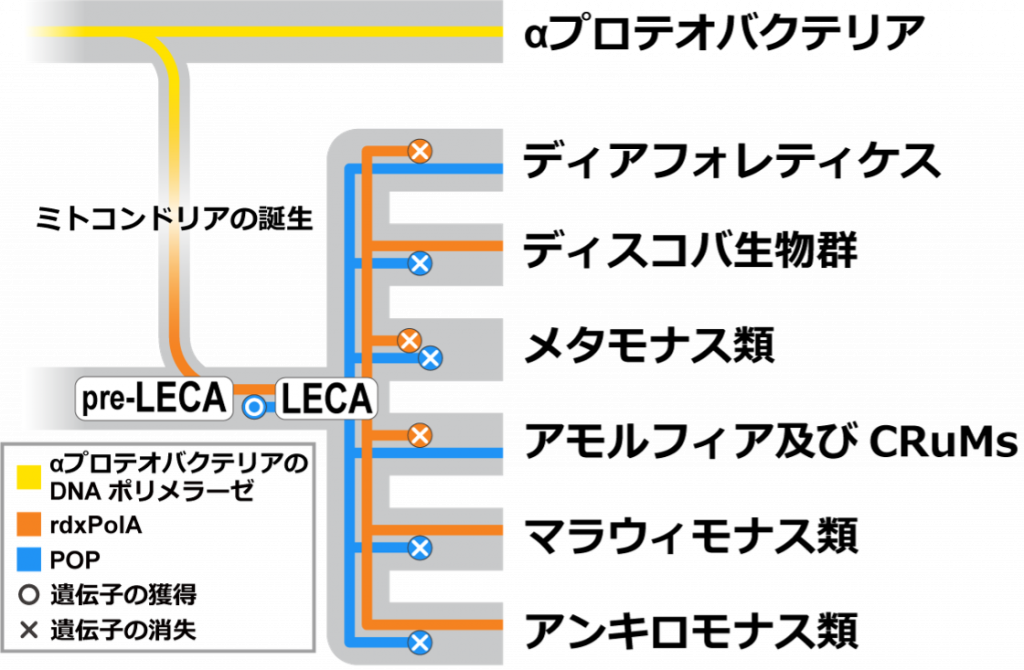
Researchers led by University of Tsukuba have discovered rdxPolA, a putative DNA polymerase involved in replicating ancestral mitochondrial genomes, in diverse eukaryotic lineages. Based on the phylogenetic distribution of rdxPolA among eukaryotes, they proposed an evolutionary scenario of DNA polymerases for mitochondrial genome maintenance in the early evolution of eukaryotes.
Tsukuba, Japan—Mitochondria are intracellular organelles that evolved from a bacterium belonging to Alpharoteobacteria, which was taken up as an endosymbiont by the common ancestor of eukaryotes. Mitochondria possess their own highly reduced genomes (known as mitochondrial genomes), which are principally the descendants of the genome of the α-proteobacterial symbiont. Phylogenetically diverse eukaryotes use a type of DNA polymerase called “POP” to maintain their mitochondrial genomes.
In this study, the researchers identified 10 novel types of DNA polymerase that are distinct from the previously known types, including POPs, across diverse eukaryotic lineages. The evolutionary origin and subcellular localization of each novel DNA polymerase were investigated. Intriguingly, one of the DNA polymerases identified in this study, rdxPolA, was found to be involved in mitochondrial DNA maintenance and is a direct descendant of the DNA polymerase in the α-proteobacterial symbiont that gave rise to the first mitochondrion. The researchers proposed a scenario for the evolution of DNA polymerases involved in mitochondrial DNA maintenance from primitive to extant eukaryotes.
These findings provide critical insights into the early evolution of the machinery for mitochondrial DNA maintenance and the establishment of mitochondria in primitive eukaryotic cells.
###
This research was supported by the Japan Society for Promotion of Sciences projects 18KK0203, 19H03280, 23H02535, and BPI05044 (to Y. Inagaki), 22J11104 (to R. Harada), and 19H03274 (to R. Kamikawa).
Professor INAGAKI Yuji
HARADA Ryo (JSPS Research Fellowship for Young Scientists DC2)
Center for Computational Sciences (CCS), University of Tsukuba
Senior Researcher YABUKI Akinori
Deep-Sea Biodiversity Research Group, Research Institute for Global Change (RIGC), Japan Agency for Marine-Earth Science and Technology (JAMSTEC)
Resercher YAZAKI Euki
Research Center for Advanced Analysis, National Agriculture and Food Research Organization
Associate Professor KAMIKAWA Ryoma
Graduate School of Agriculture, Kyoto University
第139回計算科学コロキウムを開催いたします。多数のご来聴をお待ちしております。
講演タイトル: 南半球から小惑星をレーダーで観測する
講演者: 堀内真司 博士(NASAキャンベラ深宇宙通信施設・オーストラリア連邦科学産業研究機構(CSIRO))
日時: 2024年3月8日(金) 11:00-12:00
場所: 計算科学研究センター 会議室A
言語: Japanese
要旨:
NASA Deep Space Network (DSN)の1つであるオーストラリアのキャンベラ深宇宙通信施設(Canberra Deep Space Communication Complex、CDSCC)とオーストラリアの電波望遠鏡を活用して2015年に小惑星のレーダー観測を開始し、現在までに30以上もの地球接近小惑星の検出に成功した。小惑星のレーダー観測は、それまでアメリカのGoldstoneやプエルトリコのAreciboなど、主に北半球で行われていた。しかし地球に再接近する小惑星のおよそ5%は南半球からしか観測できないので、ギャップを埋められるようになったことは、小惑星の探査・プラネタリーディフェンス・利用など、多岐にわたる分野で注目されている。講演では、レーダー観測により小惑星の軌道が精度良く決定され、また小惑星の形状や自転、表面物質の素性、表面状態などの情報が得られることを紹介し、またアストロバイオロジーへの応用の可能性についても議論する。
Keywords: 小惑星探査、電波望遠鏡、レーダー観測、NASA深宇宙局

世話人: 庄司光男
2024年2月22日
国立大学法人筑波大学
国立研究開発法人海洋研究開発機構
国立研究開発法人農業・食品産業技術総合研究機構
国立大学法人京都大学
真核生物の多様な系統から、祖先的なミトコンドリアゲノムの複製に関わるDNAポリメラーゼと考えらえるrdxPolAを発見しました。また、系統樹上でのrdxPolAの分布を検討し、真核生物初期進化から現在に至るまでの、ミトコンドリアゲノム用DNAポリメラーゼの進化シナリオを提案しました。
ミトコンドリアは、祖先真核生物の細胞内に共生した細菌(αプロテオバクテリア)から進化した細胞内小器官です。独自のゲノム(ミトコンドリアゲノム)を持っており、これはαプロテオバクテリア共生体のゲノムが縮退した結果です。真核生物の多くのグループでは、POPと呼ばれるDNA複製酵素(DNAポリメラーゼ)がミトコンドリアゲノムの複製をしています。
本研究では、真核生物の多様な系統からPOPを含めて既知タイプとは異なる10種類の新奇DNAポリメラーゼを発見しました。これらについて、それぞれの進化的起源と細胞内で機能する場所を詳細に解析した結果、その中の一つ「rdxPolA」がミトコンドリアゲノムの複製を行っており、αプロテオバクテリア共生体が持っていたDNAポリメラーゼの直系の子孫であると判明しました。rdxPolAは祖先的なミトコンドリアゲノムを複製すると考えられ、原始真核生物から、現在地球上に棲息する真核生物に至るまでの、ミトコンドリアゲノム用DNAポリメラーゼの進化シナリオが提案されました。
本研究成果は、ミトコンドリアのDNA複製機構がどのように進化してきたか、原始真核生物細胞内でミトコンドリアがどのように確立したのかを解明する上で重要な知見を提供します。
筑波大学計算科学研究センター
稲垣 祐司 教授
原田 亮(日本学術振興会特別研究員DC2)
国立研究開発法人海洋研究開発機構
矢吹 彬憲 主任研究員
国立研究開発法人農業・食品産業技術総合研究機構
矢﨑 裕規 研究員
京都大学大学院農学研究科
神川 龍馬 准教授
【掲載誌】
プレスリリース本文はこちら
日時:2024年3月6日(水) 9:00~18:00
会場:ワークショップ室
計算科学研究センター 令和5年度年次報告会を行います。
発表時間は、新任者(*印)は15分、それ以外の方は12分です(質疑応答、交代時間含む)
セッション1 (9:00-10:00 座長:堀江和正 )
9:00 朴センター長あいさつ
9:01 Kowit Hengphasatporn (生命科学研究部門・生命機能情報分野)
9:13 石塚 成人(素粒子物理研究部門)
9:25 小泉 裕康 (量子物性研究部門)
9:37 建部 修見(高性能計算システム研究部門)
9:49 吉川 耕司 (宇宙物理研究部門)
休憩 10:00-10:05
セッション2 (10:05-11:11 座長:原田隆平)
10:05 *秋山 進一郎(素粒子物理研究部門)
10:20 堀江 和正 (計算情報学研究部門・データ基盤分野)
10:32 *朝比奈 雄太(宇宙物理研究部門)
10:47 堀 優太 (生命科学研究部門・生命機能情報分野)
10:59 前島 展也(量子物性研究部門)
休憩 11:11-11:16
セッション3 (11:16-12:16 座長:秋山進一郎)
11:16 藏増 嘉伸(素粒子物理研究部門)
11:28 原田 隆平 (生命科学研究部門・生命機能情報分野)
11:40 亀田 能成 (計算情報学研究部門・計算メディア分野)
11:52 サーヴォン・ブー(計算情報学研究部門・データ基盤分野)
12:04 日下 博幸(地球環境研究部門)
昼休憩 12:16-12:36
セッション4 (12:36-13:36 座長:朝比奈雄太)
12:36 重田 育照(生命科学研究部門・生命機能情報分野)
12:48 福島 肇(宇宙物理研究部門)
13:00 Tong Xiao-Min(量子物性研究部門)
13:12 中務 孝(原子核物理研究部門)
13:24 額田 彰(高性能計算システム研究部門)
休憩 13:36-13:41
セッション5 (13:41-14:41 座長:中山卓郎)
13:41 稲垣 祐司(生命科学研究部門・分子進化分野)
13:53 萩原 聡(量子物性研究部門)
14:05 朴 泰祐(高性能計算システム研究部門)
14:17 大野 浩史(素粒子物理研究部門)
14:29 庄司 光男(生命科学研究部門・分子進化分野)
休憩 14:41-14:46
セッション6 (14:46-15:49 座長:松枝未遠)
14:46 ドアン グアン ヴァン(地球環境研究部門)
14:58 *謝 淳(計算情報学研究部門・計算メディア分野)
15:13 大須賀 健(宇宙物理研究部門)
15:25 中山 卓郎(生命科学研究部門・分子進化分野)
15:37 大谷 実(量子物性研究部門)
休憩 15:49-15:54
セッション7(15:54-16:54 座長:謝 淳)
15:54 塩川 浩昭(計算情報学研究部門・データ基盤分野)
16:06 北原 格(計算情報学研究部門・計算メディア分野)
16:18 森 正夫(宇宙物理研究部門)
16:30 佐藤 駿丞(量子物性研究部門)
16:42 藤田 典久(高性能計算システム研究部門)
休憩 16:54-16:59
セッション8(16:59-18:00 座長:藤田典久)
16:59 松枝 未遠(地球環境研究部門)
17:11 清水 則孝(原子核物理研究部門)
17:23 矢島 秀伸(宇宙物理研究部門)
17:35 天笠 俊之(計算情報学研究部門・データ基盤分野)
17:47 矢花 一浩(量子物性研究部門 )
Feb 19, 2024
Purple sulfur bacteria (PSB) convert light energy into chemical energy through photosynthesis. Interestingly, certain species can photosynthesize even in environments with low-calcium levels. Using cryo-electron microscopy, researchers from University of Tsukuba unveiled the structure of light-harvesting complexes and elucidated the mechanism that facilitates photosynthesis under low-calcium conditions.
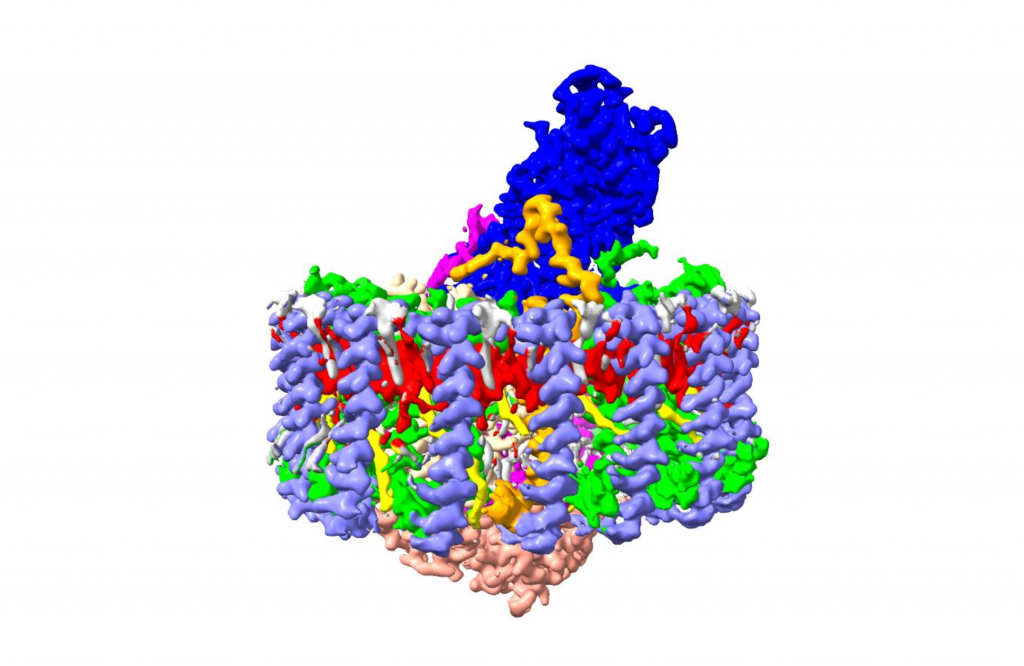
Tsukuba, Japan—Photosynthetic bacteria, unlike plants, do not generate oxygen as a photosynthetic byproduct because they use hydrogen sulfide instead of water to convert solar energy into chemical energy (electrons). This process is orchestrated by a protein complex, the light-harvesting 1-reaction center (LH1-RC). Numerous PSB thrive in calcium-rich environments, such as hot springs and seawater. In the three-dimensional LH1-RC structure, the LH1 antenna protein is typically associated with calcium. However, the photosynthetic mechanism remains elusive in Allochromatium vinosum, a model species of autotrophic bacteria capable of thriving in low-calcium or soft-water environments, as hypothetically, calcium is not involved in the photosynthetic process in this model.
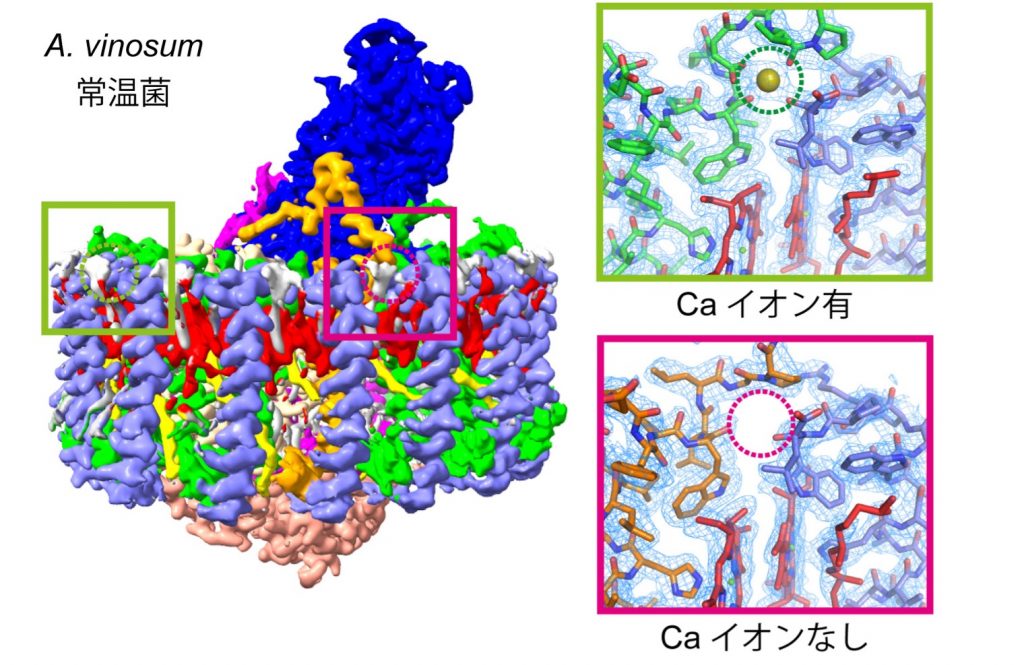
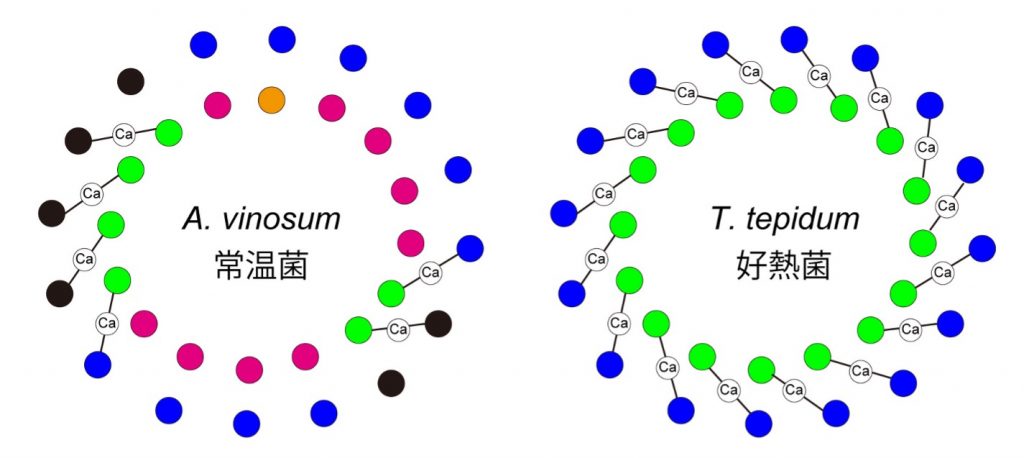
Using cryo-electron microscopy, the researchers revealed the LH1-RC structures of this model species at a resolution that enabled individual amino acid visualization. These observations revealed calcium binding only at six specific sites in the LH1 subunit. In contrast, the closely related thermophilic bacterium Thermochromatium tepidum displayed calcium attachment across all 16 LH1 subunits, indicating a calcium binding dependence on the amino acid sequence pattern. These results imply an evolutionary adaptation in this species, enabling it to bind trace amounts of calcium in low-calcium environments, thereby improving its thermal stability for photosynthesis.
These findings would potentially advance the efficient use of solar energy, and contribute to environmental protection, and highlight the capability of certain species to conduct photosynthesis in freshwater while detoxifying hydrogen sulfide, which is toxic to numerous organisms, into sulfur.
###
This research was partially supported by Platform Project for Supporting Drug Discovery and Life Science Research (Basis for Supporting Innovative Drug Discovery and Life Science Research (BINDS)) from AMED under Grant Numbers JP21am0101118 and JP21am0101116, and JP23ama121004
2024年2月19日
国立大学法人 筑波大学
紅色硫黄細菌が行う光合成では、光エネルギーを化学エネルギーに変換します。このとき、通常とは異なり、カルシウムが少ない環境でも光合成する種があります。クライオ電子顕微鏡により光を集めるタンパク質の構造を調べ、カルシウムが少なくても光合成ができるメカニズムを明らかにしました。
概要
光合成細菌が行う光合成は、酸素を発生せず、硫化水素を使って太陽光エネルギーを化学エネルギー(電子)に変換します。この役割は、タンパク質複合体であるコア光捕集反応中心複合体(LH1-RC)が担っています。中でも紅色硫黄細菌の多くは、温泉・海中といったカルシウムが豊富な環境に棲息しており、LH1-RCの立体構造では、光捕集に特化したアンテナタンパク質であるLH1にカルシウムが結合しています。しかし、カルシウム含量の少ない軟水や欠乏状態の水中でも増殖できる常温菌のモデル種アロクロマチウム・ビノサムについては、これまで、光合成にカルシウムは関与していないと考えられており、そのメカニズムは謎につつまれていました。
そこで、クライオ電子顕微鏡を用いて、本種由来のLH1-RCをアミノ酸が可視化できるレベルで観察したところ、LH1サブユニットのうち特定の6か所にのみ、カルシウムが結合していました。近縁種の好熱菌サーモクロマチウム・テピダムでは、全てのLH1サブユニット16か所にカルシウムが結合しており、アミノ酸配列のパターンに応じてカルシウムが結合できるかどうかが決まっていることが分かりました。このことから、本種は進化の過程で軟水中の微量カルシウムを結合して光合成を行うようなメカニズムを採用し、熱安定性を向上させていることが示唆されました。
本研究成果は、多くの生物にとって有毒な硫化水素を硫黄へ分解しつつ、淡水中でも光合成を行える種の、高効率な太陽光エネルギー利用への貢献や環境保全への活用が期待されます。
筑波大学 計算科学研究センター
谷 一寿 教授
光合成細菌は、植物やシアノバクテリアと異なり、光合成時に酸素を発生しないものの、非常に高い効率で太陽光エネルギーを化学エネルギー(電子)へ変換できるように進化してきました。どの光合成細菌も、植物で利用しない近赤外領域の太陽光を利用するという点では同じ特徴を持っていますが、菌の種類ごとに棲息環境が異なり、淡水から海水、温泉まで幅広く、それぞれの環境に最適な光捕集メカニズムを有しています。
酸素非発生型の光合成細菌の光合成は、進化的に古く、酸素発生型である植物の光合成に類似している部分もありますが、効率を重視した独自の進化過程を遂げたことが分かっています。特に硫化水素を使って光合成を行う紅色硫黄細菌注1)のほとんどは、熱安定性のために生育上カルシウムが必要で、温泉や海といったミネラル豊富な水中に棲息しますが、ごく一部の種ではカルシウム含量の少ない軟水にも棲息しています。このような種では、光を電子に変換するタンパク質複合体であるコア光捕集反応中心複合体(LH1-RC)注2)が、独特の進化をしていますが、その立体構造とカルシウムイオンの関係性は不明な部分が多く、カルシウムが要求されない生育環境で、高効率かつ安定的な光合成を行える仕組みは謎に包まれていました。
本研究で行った、常温菌である紅色硫黄細菌と好熱菌のLH1-RCの比較結果から、紅色硫黄細菌の高温耐性へ向けた遺伝子改変・導入を行うことで、その生物工学的な利用における効率・安定性が向上すると考えられます。また、硫化水素を含む排水処理といった環境保全にまで広く活用できると期待されます。

注1)紅色硫黄細菌
光合成細菌の仲間で、含まれるカロテノイドの種類により赤、褐色などに見える。ほとんどが嫌気性で硫黄泉、湖などの硫化水素が溜まった酸素のない環境を好んで棲息している。植物などとは異なり、光合成時には水ではなく硫化水素を使うため、酸素を発生しない。カルシウムイオンは、熱安定性に関連しているものの、硫黄代謝に直接の関連性はないため、長らく常温菌のA. vinosumは光合成にカルシウムイオンが不要だと考えられてきた。
注2)コア光捕集反応中心複合体(LH1-RC)
光エネルギーをアンテナタンパク質(LH1)で効率的に捕集し、反応中心(RC)へ伝え、光から電子への変換を行い、キノン分子により電子を伝達するタンパク質複合体。
注3)クライオ電子顕微鏡(Cryo-EM)
生体の高分子構造を立体的に解析できる手法の一種。本研究では、筑波大学生存ダイナミクスセンターのCRYO-ARM300IIと、沖縄科学技術大学院大学のTitan Krios G1を使用した。
注4)LH1サブユニット
光を集めるためのアンテナタンパク質LH1は、通常、α鎖とβ鎖と呼ばれる2種類の膜タンパク質から構成される。これらは、14~17ペアで、隙間なくリング状に並ぶことが多い。このリング状構造では、内側にα鎖が、外側にβ鎖が配列される。
注5)アイソフォーム
単一の遺伝子あるいは遺伝子ファミリーに由来している類似のタンパク質。互いに同じ機能である場合もあるが、全く異なることもある。LH1サブニットでは、α鎖とβ鎖のそれぞれが複数のアイソフォームで構成されることがある。T.tepidumは、α鎖とβ鎖ともに1種類ずつで、アイソフォームはないが、A. vinosumは、α鎖3種類とβ鎖2種類のアイソフォームが存在する。
本研究は、国立研究開発法人日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業(BINDS)JP21am0101118、 JP21am0101116、JP22am121004、科研費(JP20H05086, and JP20H02856)等の助成を受けて実施されました。
【題 名】 High-Resolution Structure and Biochemical Properties of the LH1–RC Photocomplex from the Model Purple Sulfur Bacterium, Allochromatium vinosum.
(紅色硫黄細菌のモデル種アロクロマチウム・ビノサム由来光捕集反応中心複合体の高分解能構造と生化学的特性)
【著者名】 Kazutoshi Tani, Ryo Kanno, Ayaka Harada, Yuki Kobayashi, Akane Minamino, Shinji Takenaka, Natsuki Nakamura, Xuan-Cheng Ji, Endang R. Purba, Malgorzata Hall, Long-Jiang Yu, Michael T. Madigan, Akira Mizoguchi, Kenji Iwasaki, Bruno M. Humbel, Yukihiro Kimura & Zheng-Yu Wang-Otomo
【掲載誌】 Communications Biology
【掲載日】 2024年2月12日
【DOI】 10.1038/s42003-024-05863-w
計算科学研究センター(CCS)に所属する教員・研究員の研究をわかりやすく紹介する「研究者に聞くー研究トピックス」に「vol.17『City-LES』の高速化をめぐるストーリー」を公開しました。
高性能計算システム研究部門の多田野助教の研究を紹介しています。
多田野 寛人 助教
高性能計算システム研究部門
多田野先生は、高性能計算システム研究部門の研究者です。方程式をコンピュータで速く解くための方法を研究することで、様々なモデルの計算時間短縮に貢献しています。今回は、多田野先生が都市街区気象モデル『City-LES』の高速化に携わったときの話を紹介します。
(2024.2.14 公開)
City-LESは、計算科学研究センター日下研究室で開発された、都市気象に特化したモデルです。現実の都市と同じように建物や街路樹をひとつひとつ考慮しているため、モデル内の熱放射環境は非常に複雑になっています。しかし、ラジオシティ法1)を用いれば、このような複雑な放射計算でも高精度に行うことができます。求めた放射量から地表面の温度などを算出し、LES2)モデルと結合することで、都市街区内の詳細な熱環境を再現しています。
私たちの身の回りで起こる物理現象は、偏微分方程式で記述されます。熱環境をモデルで再現するということは、偏微分方程式を差分の形で表し、連立一次方程式に書き換えて数値解を求めるということです。City-LESは解くべき方程式の数が非常に多いため、計算に長い時間がかかり、この部分が高速化のボトルネックになっていました。開発者の日下先生からこのことを相談された多田野先生は、面白そうだとCity-LES開発に協力することを決めました。

図1:City-LESによってシミュレートされた東京駅周辺の地上気温分布(出典:筑波大学日下研究室)
いきなりですが、頭の体操です。
![]()
を解いてみましょう。解けましたか?

このように、変数を1つずつ消去して、解を求める方法を「直接解法」と呼びます。この問題は変数がxとyの2つだったので、操作は2回で済みました。では、変数が数千個、数万個ならどうしますか? 数千回、数万回と操作を繰り返すのは大変です。こうした場合は、連立一次方程式を行列の形に表して解くことを考えます。具体的には、連立一次方程式を次のように変形します。
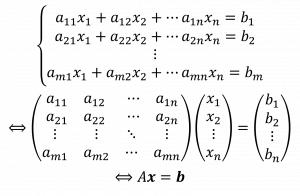
ここで、Aは係数行列、xは未知ベクトル、bは既知ベクトルです。つまり、連立一次方程式を解くということは、xを解として求めることにほかなりません。直接解法で解くのは大変なので、こういう場合は「反復解法」を使います。反復解法とは、はじめにxの解を仮定して、その値を更新していくことにより、真の解に近づけていく方法です。反復解法は1回当たりの演算量が少ないため、反復回数が少ない場合には直接解法よりも計算時間が短くなります。つまり、計算時間短縮の可能性を秘めているのは、反復解法ということになります。
多田野先生は、反復解法のひとつである「クリロフ部分空間反復法」を採用し、計算時間短縮に挑むことにしました。クリロフ部分空間反復法の概念図を図2に示します。最初に仮定する解がx0で、x0, x1, …, xkと解が更新されるにつれて、真の解xに近づいていることが分かります。解の更新の仕方にはいくつもの種類があり、今回はそのうち3つの方法(CG法、CR法、Orthomin法)で計算速度を比較することにしました。
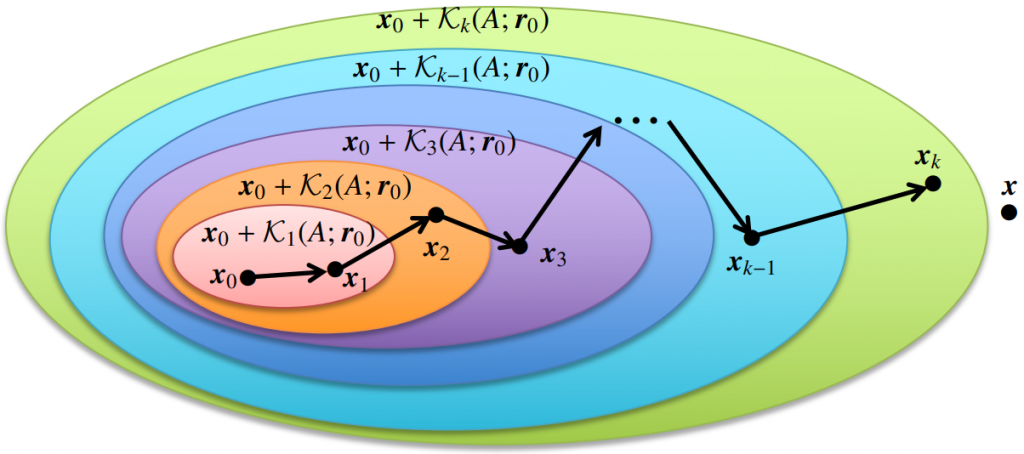
図2:クリロフ部分空間反復法の概念図。行列 と非ゼロベクトル から生成されるベクトル列で張られる部分空間:Kk(A, v)=span(v, Av, …, Ak-1v)をクリロフ部分空間という。クリロフ部分空間を用いて連立一次方程式Ax=bの近似解を生成する方法をクリロフ部分空間反復法という。
反復解法では、反復回数を減らすために「前処理」を行うことがよくあります。前処理の方法には色々ありますが、多田野先生が目をつけたのは「マルチグリッド法」です。マルチグリッド法では、グリッドサイズを一気に大きくして大雑把な解を求めた後、徐々にグリッドサイズをもとに戻していき、詳細な解を求めていきます(図3)。解に見当をつけながら解くことになるので、効率的な解法だと言われています。
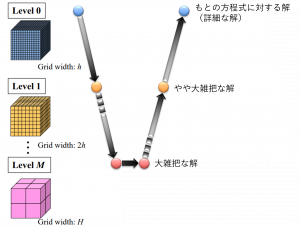
図3:マルチグリッド法の概念図
そこで、CG法、CR法、Orthomin法という3つの反復解法について、マルチグリッド前処理により計算時間や反復回数がどう変わるかを調べました。その結果を図4に示します。まず、前処理を行った場合(青色、緑色)と前処理を行わなかった場合(赤色)で比較すると、3つの反復解法すべてで前処理を行った方が計算時間がかなり短くなっています(図4a)。また、前処理を倍精度3)で行った場合(青色)と単精度3)で行った場合(緑色)を比較すると、単精度で行った場合の方が計算時間が短くなっています(図4a)。倍精度の方が精度は高くなりますが、今回の前処理の計算では“計算が高速化できる”というメリットの方が大きいため、計算時間が最も短かった「単精度マルチグリッド前処理付きOrthomin法(黄色枠)」が最適な手法と言えそうです。図4bによると、このときの平均反復回数は1.03回となっています。これは、ほとんど1回の更新だけで真の解が求められた、つまり、アルゴリズム的には計算速度が最大に達したことを意味します。
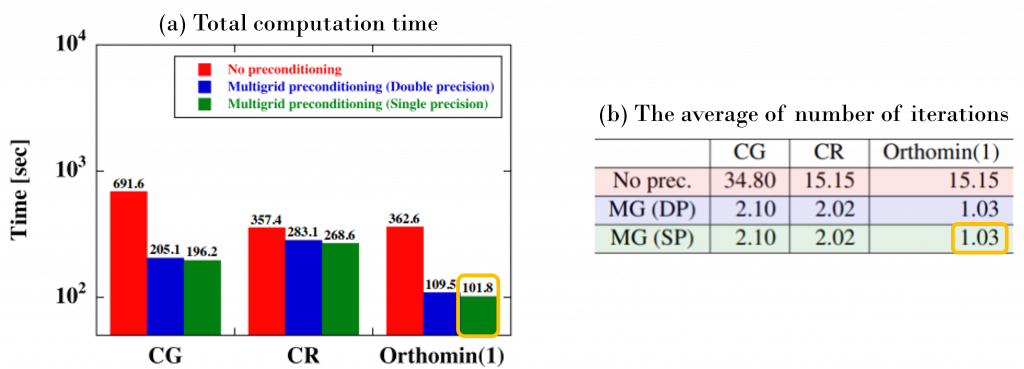
図4:CG法、CR法、Orthomin法に対するマルチグリッド前処理の効果。101本の線形方程式を解いたときの (a) 総計算時間、及び、(b) 平均反復回数。赤色:前処理なし、青色:倍精度でのマルチグリッド前処理あり、緑色:単精度でのマルチグリッド前処理あり。
多田野先生がCity-LESの高速化で大成功を収めることができたのは、“先入観にとらわれず、まずは試しにやってみる”というポリシーで研究されていたためです。― やはり、自分の編み出した計算を速くするテクニックが、実際のアプリケーションに役立てられると嬉しい。今後は素粒子物理学や量子力学など、様々な分野のアプリケーション開発にも積極的に携わりたい ― 多田野先生の技術を欲している研究者は、ほかにもいるはずです。「多田野先生のおかげでアプリケーションが高速化した」というニュースが、また飛び込んでくることを期待しています。
(文・広報サポーター 松山理歩)
第138回計算科学コロキウムを開催いたします。多数のご来聴をお待ちしております。
講演タイトル: 太陽系外惑星における光合成シグナルの観測可能性について
―惑星の輻射環境と光合成色素の光物性―
講演者:小松 勇 博士(自然科学研究機構アストロバイオロジーセンター・国立天文台)
日時: 2024年2月22日(木) 13:00-14:00
場所: 計算科学研究センター 会議室C
言語: Japanese
要旨:
将来の太陽系外惑星観測において光合成由来のシグナルを検出する可能性を探るために天文学・地球科学・化学・生物学などの複数分野に跨った議論が要求されており、計算科学による貢献が期待される。本発表では惑星における放射伝達を考慮したモデル計算によって、光合成由来の蛍光が将来どのように観測され得るかについて調べた研究について紹介する。また、量子化学計算を用いた惑星で実現可能な色素探索の研究も発表する。
Keywords:アストロバイオロジー、光合成、太陽系外惑星、放射伝達、量子化学計算

世話人: 庄司光男
筑波大学計算科学研究センターは、文部科学省の令和5年度 共同利用・共同研究システム形成事業「学際領域展開ハブ形成プログラム」において「AI 時代における計算科学の社会実装を実現する学際ハブ拠点」として採択されました。本事業では、スーパーコンピュータと計算科学手法を用いた問題解決において、産官学の連携による新たな需要の開拓と、企業における計算科学手法の導入の裾野を広げることを大きな目的としています。拠点としての活動の活性化と分野を跨いだ知の共有を目的として、キックオフシンポジウムを以下の通り開催致します。
皆様奮ってご参加ください。
日時: 2024年3月13日(水)13:00-17:30
場所: 筑波大学 計算科学研究センター ワークショップ室 / オンライン(Zoom)
参加登録: 参加登録は締切ました
* 参加登録をされた方にZoom URL等の情報をお送りしました。メールが届いていない方は、下記お問い合わせ先までご連絡ください。
参加費: 無料(ただし懇親会参加の場合は3,000円)
| タイトル | 講演者 | |
| 13:00 – 13:15 | 開会のあいさつ | 朴 泰祐 (筑波大学計算科学研究センター センター長) |
| 13:15 – 13:45 | 都市街区気象モデル City-LESの概要と暑さ指数分布の再現精度 | 日下 博幸 (筑波大学計算科学研究センター) |
|
13:45 – 14:15 |
City-LESによる都市気象予測の実利用への取り組み | 池田 亮作・坂本 晃平 (株式会社ウェザーニューズ) |
| 14:15 – 14:50 | 計算科学による表面・界面の化学反応・デバイス動作過程の微視的理解と制御 | 大谷 実 (筑波大学計算科学研究センター) |
| 14:50 – 15:15 | 材料データベースの統計解析による新奇触媒材料創生の取り組み | 井部 将也 (トヨタ自動車先端材料技術部) |
| 15:15 – 15:30 | 休憩(15分) | |
| 15:30 – 16:00 | 計算メディカルサイエンス生体分子医科学分野:医薬分野との共同 | 重田 育照 (筑波大学計算科学研究センター) |
| 16:00 – 16:30 | 学際研究で実現する高精度な環状ペプチド分子シミュレーション | 秋山 泰 (アヘッド・バイオコンピューティング株式会社 取締役CTO) |
| 16:30 – 17:25 | パネルディスカッション | 朴 泰祐 日下 博幸 大谷 実 重田 育照 坂本 晃平 井部 将也 秋山 泰 |
| 17:25 – 17:30 | 閉会のあいさつ | 重田 育照 (筑波大学計算科学研究センター・筑波大学 研究担当副学長) |
問い合わせ:pr[at]ccs.tsukuba.ac.jp ([at]→@に変換ください)
計算科学研究センター(CCS)に所属する教員・研究員の研究をわかりやすく紹介する「研究者に聞くー研究トピックス」に「vol.16 素粒子理論にテンソルネットワークで挑む」を公開しました。
素粒子物理研究部門の秋山助教の研究を紹介しています。
秋山進一郎 助教
秋山先生は素粒子の振る舞いを記述する枠組みの一つである格子上の場の理論の研究をされている先生です。
膨大な計算コストがかかる素粒子理論の計算に、テンソルネットワークという新しい手法を使って挑戦しようとしています。
(2024.1.25 公開)
この世界の最小構成単位だと考えられている素粒子は「素粒子標準模型」という理論によって素粒子同士の間でどのような力が働くか記述されています。この素粒子標準模型を用いればこの世界にある原子や分子の構造や、宇宙の成り立ちなどが理解できることになります。
素粒子標準模型は量子色力学(QCD)という理論を含んでいます。QCDはクォークとグルーオンと呼ばれる素粒子を記述する理論で、クォーク間で働く力はクォーク間の距離が長くなるほど強くなるという性質があります(高校で習う電磁気学では、電子間で働く力は電子間の距離が長くなるほど弱くなります)。このような性質がある理論は中々手で解くことができません。そこで、QCDによって記述される物理現象を調べるには、スーパーコンピュータを使った数値シミュレーションが非常に重要になってきます。特に、「格子QCD」と呼ばれる分野では、私たちの住む世界を一旦メッシュ状(格子)に区切って、その上でQCDに基づく計算を色々と行います(格子QCDだと摂動計算(注1)という近似をしなくて済むという利点があります)。
この格子QCDで従来使われてきた方法が「モンテカルロ法」です。モンテカルロ法は乱数を使った数値計算手法の総称であり、積分を数値的に計算する上で重宝される手法の一つです。格子QCDではクォークやそれらの間で働く力を伝えるグルーオンについての膨大な積分を解かないといけません。こうした積分をコンピュータで計算する場合、高校数学で習う区分求積法のような考え方を使ってしまうと、計算量が大きすぎてスーパーコンピュータをもってしても太刀打ちできません。一方、モンテカルロ法を使うと「積分結果への寄与が大きな積分領域になるべく注目する」ことで計算量を大幅に減らすことができます。具体的には、被積分関数を確率とみなし、その確率が高い点を重点的にサンプルすることで計算量を抑えます。
ところが、モンテカルロ法はいつでも使えるわけではありません。例えば、被積分関数が複素数になってしまう積分は扱えません。なぜなら、複素数の値は確率と見なすことができないからです。この問題はモンテカルロ法の「符号問題」と呼ばれ、素粒子理論の研究だけでなく、物理学の様々な分野でよく現れる問題です。どんなに興味深い物理現象があっても、符号問題が生じてしまうと、その現象を基礎理論の立場から解明することが極めて困難になってしまいます。素粒子の分野では、例えば中性子星(注2)の中の状態などを調べるために必要なQCDの計算でこの符号問題が現れます。そのため、QCDの立場から中性子星の内部状態を理解することは未だにできていません。
そこで、秋山先生が注目したのが「テンソルネットワーク法」です。
テンソルネットワーク法では、計算したい対象を「テンソル」と呼ばれる行列を一般化したものを使って表現しなおします。例えば、先ほどの積分の場合だと、解きたい積分を多数のテンソルが組み合わさったもの(ネットワーク)に書き換えます。このように問題を書き換えることで、これまでの方法(例えばモンテカルロ法)とは全く違う切り口から問題にアプローチすることができるのです。
しかし、単に問題を書き換えただけでは元々の問題が持っていた難しさを必ずしも解決できるとは限りません。実際、テンソルネットワーク法は時空間次元が1次元や2次元の問題では広く使われていましたが、高次元への応用例は少なく、特に4次元(私たちの住む世界は時間1次元、空間3次元の4次元世界です)の問題への応用は前例がありませんでした。
そこで、秋山先生はスーパーコンピュータの利用を前提として4次元系向けのテンソルネットワーク法の数値計算コードを設計・実装し、世界で初めて4次元時空上の理論をテンソルネットワーク法で計算することに成功しました。また、秋山先生は一度テンソルネットワークで書き換えられた問題をさらに別のテンソルネットワークで表現することで、計算量をさらに抑える手法を開発しました(関連論文はこちら)。さらに、4次元系に対するテンソルネットワーク法のテクニックを応用して、QCDにも応用可能な新しいテンソルネットワーク手法の研究を進めています(図)。今後はGPU(注3)を用いたり、機械学習の手法も組み込んだりすることで、より計算の効率化を図り、素粒子物理学における様々な問題をテンソルネットワーク法で精密に計算できるようにしたいと考えています。
(文・広報サポーター 類家千怜)
1)摂動計算:解きたい問題を、すでに解の存在する部分と残りの部分に分けて、残りの部分の影響が小さいと近似して問題を解くテクニックのこと。
2)中性子星:質量の大きな恒星の最終形態であり、密度は太陽の1014倍以上の天体。中性子が主な構成要素になっている。
3)GPU:Graphics Processing Unitの略。本来PCサーバにおけるグラフィックス処理を目的として作られた専用プロセッサだが、近年はその高い演算性能で高性能計算へ転用されている。